在4種主要免疫細胞類型中,細胞背景對miR-155介導基因調控的影響
許多microRNA及其靶mRNA在不同細胞類型中共表達。然而,尚不清楚它們是以獨立于細胞環境還是依賴細胞環境的方式受到調節。近日,來自美國的Rudensky和他的團隊在Nat Immunol(IF=21.809)在線發表了題為“The effect of cellular context on miR-155-mediated gene regulation in four major immune cell types”的文章。探索了miR-155的轉錄組范圍靶向和基因調控,其活化誘導的表達在先天性和適應性免疫中起重要作用。在激活的miR-155充足或者缺陷的巨噬細胞,樹突狀細胞和T和B淋巴細胞中,通過不同的iCLIP定位miR-155靶標,用RNA-seq進行mRNA定量,并用的poly(A)-seq進行3'非翻譯區(UTR)的分析,他們鑒定了miR-155差異結合的許多靶標。盡管選擇性切割和多腺苷酸化(ApA)有助于不同的miR-155與一些轉錄物的結合,但在大多數情況下,相同的3'-UTR同源異構蛋白在細胞類型中被差異調節,因此表明不依賴ApA和細胞環境依賴性miR-155介導的基因調控。他們的研究提供了miR-155調控網絡的綜合圖譜,并為關鍵免疫細胞類型中的背景依賴性和背景獨立的miRNA介導的基因調控提供了寶貴的資源。
技術路線
主要結果
1 mir -155介導的調控是環境特異的。
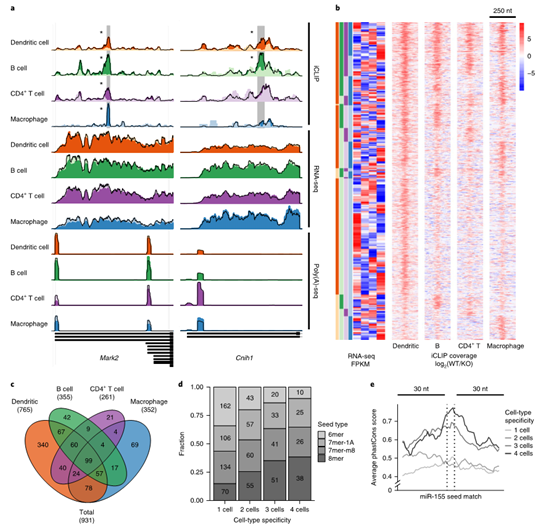
圖1 miR-155介導的Ago結合發生在四種免疫細胞類型的不同位點。
a,四種細胞類型中miR-155普遍結合位點與差異結合位點的實例。 iCLIP,RNA-seq和poly(A)-seq文庫的標準化讀長覆蓋率,野生型(WT)為深色軌跡,miR-155-敲除(KO)為淺色軌跡。含有miR-155-seed的iCLIP峰以灰色矩形突出顯示;星號表示WT和KO覆蓋范圍之間的顯著差異(錯誤發現率(FDR)<2.5%)。 b,共表達基因中miR-155依賴性位點的總結,包括用不同iCLIP鑒定的3'-UTR,CDS和5'-UTR位點。每行代表miR-155六聚體種子匹配周圍的250bp;顏色表示歸一化WT與miR-155-KO iCLIP覆蓋率比值,并取2為底數的對數。含有miR-155位點的相同基因的RNA表達在熱圖中并排顯示(WT RNA-seq log10FPKM,通過行標準化)。根據四種細胞類型中的特異性結合對位點進行分類,并且通過對相應基因的RNA-seq FPKM值的層次聚類來確定每個類別內的順序。 c,共表達基因中miR-155依賴性iCLIP位點的維恩圖。 d,共表達基因中miR-155依賴性位點的種子類型組成。 e,共表達基因中miR-155依賴性位點的PhastCons評分(對于小鼠和39種其他胎盤哺乳動物之間的多基因組比對)。顯示了來自獨立iCLIP(n = 4),RNA-seq(n = 3)和poly(A)-seq(n = 4)實驗的數據的分析。
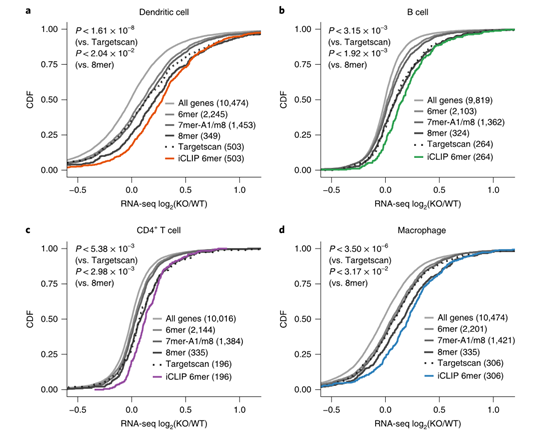
圖2 miR-155抑制四種免疫細胞類型中不同的基因集合。
a-d,在樹突細胞(a)B細胞(b),CD4+ T細胞(c)和巨噬細胞(d)中,用不同基因集合的累積分布函數(CDF)顯示miR-155-KO和WT細胞之間RNA-seq表達變化的分布。基因集合包括所有表達的基因,具有3'-UTR miR-155六聚體/七聚體-A1 /七聚體-m8 /八聚體-種子匹配的基因和含有具有六聚體種子匹配的3'-UTR miR-155依賴性iCLIP位點的基因(FDR <2.5%)。顯示利用Targetscan 7.0預測的最高context++得分的miR-155-靶基因(與通過不同的iCLIP鑒定的miR-155-靶基因數目相同)。P值通過單側Kolmogorov-Smirnov檢驗測試確定。數據代表獨立的iCLIP(n = 4)和RNA-seq(n = 3)實驗。
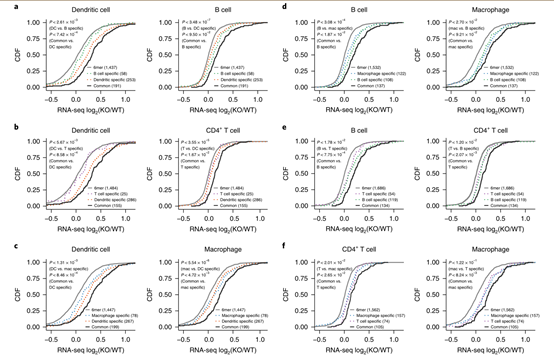
圖3特定背景下的miR-155靶向作用導致不同細胞類型之間基因調節的差異。
a-f,四種免疫細胞的六個成對比較(如圖所示),描繪了含有共同(實線)和細胞類型特異性(虛線)3'-UTR miR-155依賴性iCLIP位點的基因的去阻遏,以CDF的形式顯示。具有3'-UTR miR-155-種子匹配的基因為參考。每個成對比較中僅包括共表達的基因(WT RNA-seq FPKM> 1且差異<16倍)。在每個圖中,顯示了來自單側KS測試的兩個P值。第一次KS測試對應于在該細胞類型中鑒定的所有miR-155靶基因與僅在其他細胞中靶向的基因之間的比較,第二次對應于共同靶基因和對該細胞類型特異的靶基因之間的比較。顯示了四個獨立的iCLIP和三個獨立的RNA-seq實驗的結果。 DC,樹突狀細胞; mac,巨噬細胞。

圖4 細胞類型依賴性miR-155介導抑制的驗證。
報告基因攜帶了表現出環境特異靶向性基因的3'-UTR,在B細胞和樹突細胞中表達。顯示了Hif1a和Jarid2(優先在B細胞中被抑制),Actr10和Terf1(B細胞特異性靶標)和Tbca,Uqcrfs1和Zfp277(樹突細胞特異性靶標)的結果。折疊抑制被定義為3'-UTR報告基因的突變體和野生型的標準化熒光素酶活性的比率。箱形圖顯示匯總統計數據,包括中位數,第25和75百分位數,1.5倍四分位數間距(IQR)和異常值。誤差棒,s.e。 來自至少三個生物學上獨立的樣品(對于Hif1a,Jarid2,Tbca和Uqcrfs1,n = 3;對于Actr10,Terf1和Zfp277,n = 4); P值通過雙側t檢驗確定。 * P <0.05; ** P <0.01
2 內源性RNA競爭不太可能影響miR-155靶向,而ApA對細胞類型特異性miR-155靶向的貢獻有限。
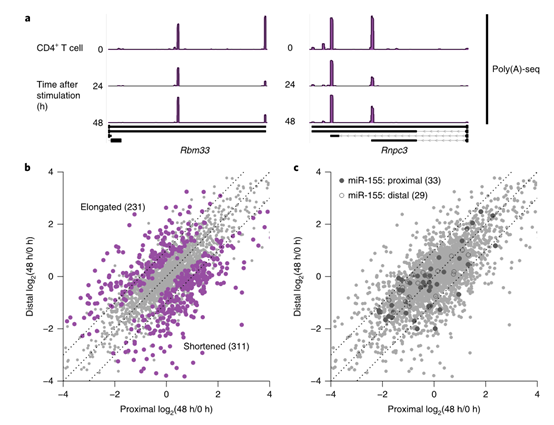
圖5 Poly(A)-seq捕獲CD4+ T細胞活化期間3'-uTR-同源異構蛋白使用的變化。
a,在CD4+ T細胞活化期間,同源異構蛋白使用中具有顯著(FDR <5%)變化的的兩個3'-UTR實例。軌跡表示激活后0小時,24小時和48小時的歸一化poly(A)-seq讀長覆蓋率。b,在CD4 + T細胞活化后48小時,具有兩種主要同源異構蛋白的3'-UTR的3'-UTR-同源異構蛋白使用的變化。突出顯示的基因在3'-UTR使用中顯示出顯著的(FDR <5%)變化。c,如在b中,但突出顯示了含有miR-155靶位點的雙同源異構蛋白的3'-UTR。突出顯示了含有近端(實心)和遠端(空心)miR-155-靶位點的3'-UTR。顯示了三個獨立的poly(A)-seq實驗的結果。
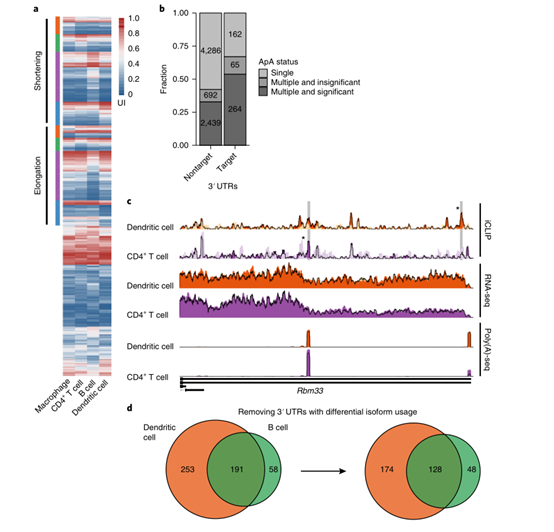
圖6 替代聚腺苷酸在miR-155調控細胞-環境依賴性基因表達中的作用。
a. multi-isoform 3′UTR在所有四個細胞類型熱圖變化。b、3′UTR包含mir - 155細胞類型特異的ApA目標和顯示差異。c、iCLIP、RNA-seq和poly(A)-seq在Rbm33的樹突狀細胞和CD4 + T細胞中的讀取-覆蓋。d, Venn圖顯示了樹突狀細胞和B細胞中共享和細胞特異性mir -155靶基因的數量,這些基因在去除具有細胞類型特異性ApA差異的基因之前和之后變化。
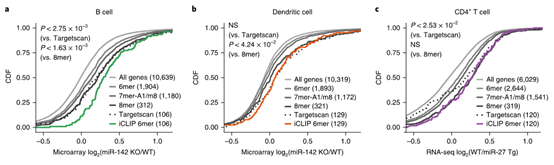
圖7 其他miRNA的iCLIP靶位點表達誘導顯著的基因抑制。
a-c,具有miR-142a KO的B細胞(a)和樹突細胞(b)以及具有miR-27a過表達(c)的CD4 + T細胞中的mRNA表達變化,顯示為不同基因組的CDF。






